使用RepeatMasker屏蔽基因组重复序列
简介
复杂的基因组大部分是可转座元件和其他重复序列,在全基因组比对或者基因注释之前需要屏蔽重复序列,否则就会浪费计算资源,也会得到许多无意义的结果。
RepeatMasker(官网:http://www.repeatmasker.org/RMDownload.html)是一款识别基因组中重复序列和低复杂度序列的软件。RepeatMasker依赖序列搜索引擎(HMMER、Cross_Match、ABBlast或者RMBlast)和重复序列数据库(Dfam和Repbase)来屏蔽重复序列。
对于非模式生物,这两个数据库覆盖有限,因此需要先使用RepeatModeler从头预测重复序列,然后将RepeatModeler预测的序列作为RepeatMasker的输入,来完成最终的重复序列屏蔽。更多的信息请访问RepeatMasker的官网http://www.repeatmasker.org/RepeatModeler/。
安装依赖
本文使用RepeatMasker-4.1.2-p1,它需要以下依赖:
- perl 5.8.0或者更高的版本
- Python 3和h5py库
- 序列搜索引擎
- TRF - Tandem Repeat Finder
- 重复序列数据库
我的HOME路径是/home/chenwen/,软件安装都安装在/home/chenwen/software/,请记得修改为适合自己的路径。
RepeatMasker支持序列4种搜索引擎,我们没有必要全都装上,只安装RMBlast也可以。
安装Cross_Match
按照官网http://www.phrap.org/说明,使用学术机构的邮箱申请软件。申请通过后,软件通过邮件发送给我们。这里提供我的副本phrap_cross_match_swat_1.090518.zip。
1 | cd ~/software/ |
安装RMBlast
官网http://www.repeatmasker.org/RMBlast.html
1 | cd ~/software/ |
安装HMMER
官网http://hmmer.org/,RepeatMasker官方推荐使用HMMER v3.2.1,而不是其他版本。
1 | cd ~/ |
安装ABBlast
在http://blast.advbiocomp.com/licensing/填写表格后,软件和使用许可通过邮件发送给我们。ABBlast解压即可用,使用许可license.xml需要放置到~/.config/ab-blast目录下。
1 | cd ~/software/ |
安装trf
官网https://github.com/Benson-Genomics-Lab/TRF
1 | cd ~/software/ |
安装和配置RepeatMasker
下载RepeatMasker
1 | cd ~/software/ |
下载RepBase数据库
1 | cd ~/software/RepeatMasker/ |
更新Dfam数据库
RepeatMasker带一个小的Dfam数据库,我们需要重新下载完整版的数据库。解压后的Dfam.h5文件83G,非常大。
1 | cd ~/software/RepeatMasker/Libraries/ |
安装h5py
1 | pip install h5py |
配置RepeatMasker
1 | cd ~/software/RepeatMasker/ |
配置过程中,必须配置trf路径,4种搜索引擎的路径可以选择配置。
1 | TRF_PRGM: /home/chenwen/software/trf |
如果配置成功,将有如下提示。
配置成功之后,删掉巨大的Dfam.h5文件。
1 | rm ~/software/RepeatMasker/Libraries/Dfam.h5 |
将RepeatMasker添加到环境变量
1 | echo "export PATH=$HOME/software/RepeatMasker:\$PATH" >> ~/.bashrc |
运行RepeatMasker
这里我们以从Ensembl下载的斑马鱼基因组为例。
1 | mkdir ~/test_RepeatMasker |
1 | RepeatMasker Danio_rerio.fa -species "Danio rerio" -e rmblast -xsmall -s -gff -pa 12 |
参数说明
1 | -e rmblast 指定搜索引擎为rmblast,还可以选择crossmatch、abblast或者hmmer |
结果解读
斑马鱼基因组大小约1.4G,上述命令在我的i5 10400(6核12线程)电脑上面跑了约10.5小时。结果文件主要有:Danio_rerio.fa.tbl、Danio_rerio.fa.masked、Danio_rerio.fa.out、Danio_rerio.fa.out.gff、Danio_rerio.fa.cat.gz。.cat.gz是重复序列与基因组序列的比对文件,其他文件我们打开看一下。
.tbl文件是重复序列的统计信息,我们可以看到斑马鱼有60%以上的重复序列。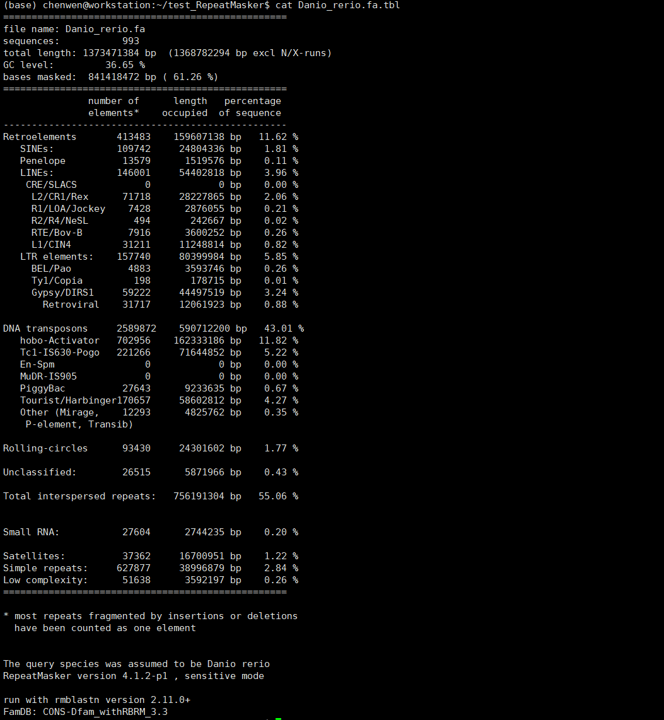
.masked文件是屏蔽重复序列后的基因组文件,这里是软屏蔽,重复序列用小写字母表示。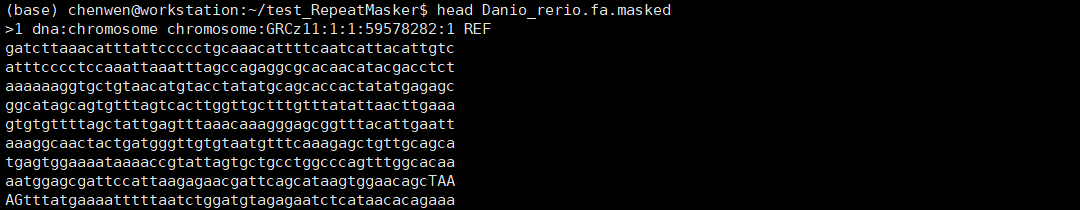
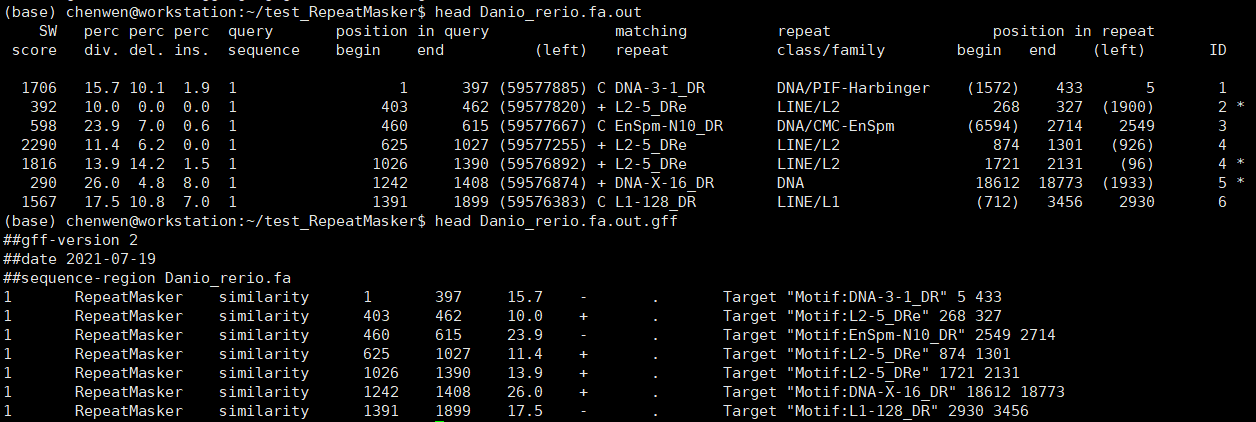
.out文件是RepeatMasker默认的输出结果,.out.gff文件是相应的GFF文件。