ZFLNCG08739
Basic Information
Chromesome: chr15
Start: 45698953
End: 45699561
Transcript: ZFLNCT13478 ZFLNCT13479 ZFLNCT13480 ZFLNCT13481 ZFLNCT13482 ZFLNCT13483 ZFLNCT13484 ZFLNCT13485 ZFLNCT13486
Known as:
Genome Browser
Express profile
Download
Express in top 10 samples
| Sample | Tissue | Condition | FPKM |
|---|---|---|---|
| SRR527832 | 16-36 hpf | normal | 298.83 |
| SRR1104058 | heart | normal | 269.11 |
| SRR516123 | skin | male and 3.5 year | 246.95 |
| SRR038625 | embryo | traf6 morpholino | 246.71 |
| SRR658543 | 6 somite | Gata5 morphan | 245.32 |
| SRR749515 | 24 hpf | eif3ha morphant | 244.08 |
| SRR941753 | posterior pectoral fin | normal | 243.05 |
| SRR658547 | 6 somite | Gata5/6 morphant | 233.41 |
| SRR1035981 | 13 hpf | rx3+/+ or rx3+/- | 232.88 |
| SRR038627 | embryo | control morpholino | 230.82 |
Express in tissues
Correlated coding gene
Download
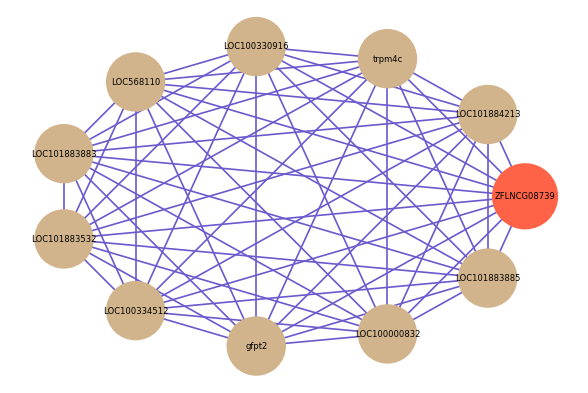
| gene | correlation coefficent |
|---|---|
| LOC101884213 | 0.73 |
| trpm4c | 0.71 |
| LOC100330916 | 0.70 |
| LOC568110 | 0.70 |
| LOC101883883 | 0.69 |
| LOC101883532 | 0.69 |
| LOC100334512 | 0.68 |
| gfpt2 | 0.68 |
| LOC100000832 | 0.68 |
| LOC101883885 | 0.68 |
Gene Ontology
Download
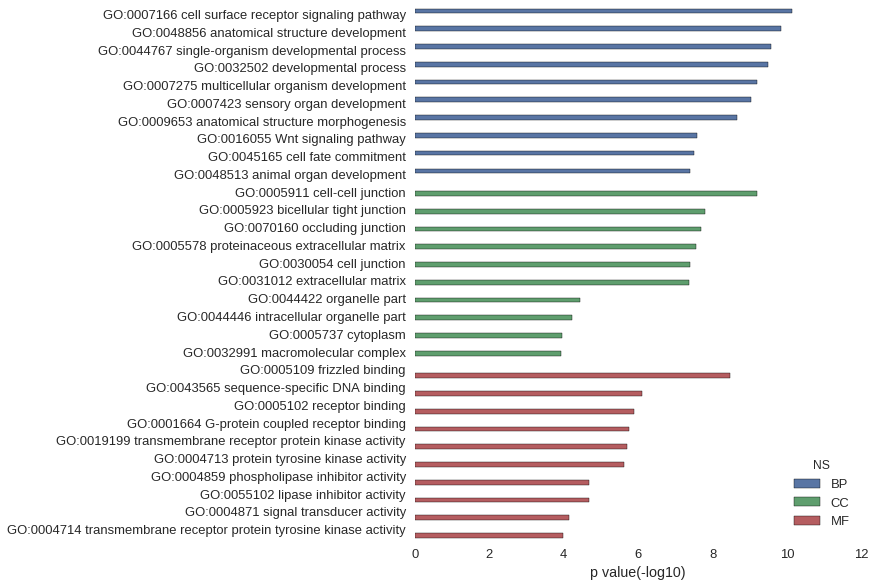
| GO | P value |
|---|---|
| GO:0007166 | 7.21e-11 |
| GO:0048856 | 1.46e-10 |
| GO:0044767 | 2.70e-10 |
| GO:0032502 | 3.13e-10 |
| GO:0007275 | 6.55e-10 |
| GO:0007423 | 9.09e-10 |
| GO:0009653 | 2.18e-09 |
| GO:0016055 | 2.56e-08 |
| GO:0045165 | 3.08e-08 |
| GO:0048513 | 3.97e-08 |
| GO:0005911 | 6.24e-10 |
| GO:0005923 | 1.58e-08 |
| GO:0070160 | 1.99e-08 |
| GO:0005578 | 2.79e-08 |
| GO:0030054 | 4.14e-08 |
| GO:0031012 | 4.19e-08 |
| GO:0044422 | 3.55e-05 |
| GO:0044446 | 6.14e-05 |
| GO:0005737 | 1.14e-04 |
| GO:0032991 | 1.22e-04 |
| GO:0005109 | 3.31e-09 |
| GO:0043565 | 7.86e-07 |
| GO:0005102 | 1.28e-06 |
| GO:0001664 | 1.75e-06 |
| GO:0019199 | 2.00e-06 |
| GO:0004713 | 2.38e-06 |
| GO:0004859 | 2.07e-05 |
| GO:0055102 | 2.07e-05 |
| GO:0004871 | 7.06e-05 |
| GO:0004714 | 1.07e-04 |
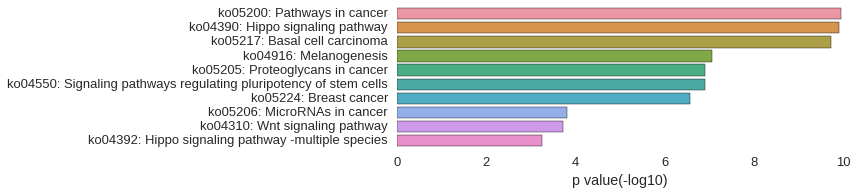
KEGG Pathway
Download
Conservation
OMIM
OMIM of this lncRNA gene hava does not exist!
Sequence Download
>ZFLNCG08739
AAATGTTTACCTCATCACTGCTCGATGACTTGCGTCTACGATGATGTCCACGTCCGTGTCCATGGCCATG
ATGGCGCTCCTGTCCGTGATGATGTCCAAATCCGCGTTGGTGTCCGTGTTCATATCCATATCCGGGTCCT
TGAGGTTGTCCATATCCAGGTCCTTGAGGTTGTCCGTATCCAGGTCCTTGAGGTTGTCCGTATCCAGGTC
CTTGAGGTTGTCCATATCCAGGTCCCTGAGGTTGTCCATATCCGGGTCCTTGAGGTTGTCCATATCCGGG
TCCTTGAGGTTGTCCATATCCGGGTCCTTGAGGTTGTCCATATCCAGGTCCTTGAGGTTGTCCATATCCA
GGTCCTTGAGGTGGTCCATAACCAGGTCCTTGAGGTGGTCCATATCCAGGTCCTTGAGGAACTCCGGGTC
CATATCCAGGTCCTTGAGGCACTCCAGATGCTGGTCCATATCCGGGTCCTTGAGGCGCTACGGGTCCTGG
TCCATACGCGGGTCCTTGAGGCACACCAGGTGCAGGTCCATATCCAGGTCCTTGAGGCAATACGGGTCCG
GGTCCTTGAGGCACTACAGGTCCAGGTCCATATGCAGGTCCTTGACCC
AAATGTTTACCTCATCACTGCTCGATGACTTGCGTCTACGATGATGTCCACGTCCGTGTCCATGGCCATG
ATGGCGCTCCTGTCCGTGATGATGTCCAAATCCGCGTTGGTGTCCGTGTTCATATCCATATCCGGGTCCT
TGAGGTTGTCCATATCCAGGTCCTTGAGGTTGTCCGTATCCAGGTCCTTGAGGTTGTCCGTATCCAGGTC
CTTGAGGTTGTCCATATCCAGGTCCCTGAGGTTGTCCATATCCGGGTCCTTGAGGTTGTCCATATCCGGG
TCCTTGAGGTTGTCCATATCCGGGTCCTTGAGGTTGTCCATATCCAGGTCCTTGAGGTTGTCCATATCCA
GGTCCTTGAGGTGGTCCATAACCAGGTCCTTGAGGTGGTCCATATCCAGGTCCTTGAGGAACTCCGGGTC
CATATCCAGGTCCTTGAGGCACTCCAGATGCTGGTCCATATCCGGGTCCTTGAGGCGCTACGGGTCCTGG
TCCATACGCGGGTCCTTGAGGCACACCAGGTGCAGGTCCATATCCAGGTCCTTGAGGCAATACGGGTCCG
GGTCCTTGAGGCACTACAGGTCCAGGTCCATATGCAGGTCCTTGACCC