ZFLNCG13245
Basic Information
Chromesome: chr24
Start: 43872233
End: 43873224
Transcript: ZFLNCT20554 ZFLNCT20555 ZFLNCT20556 ZFLNCT20557 ZFLNCT20558 ZFLNCT20559 ZFLNCT20560 ZFLNCT20561 ZFLNCT20562
Known as:
Genome Browser
Express profile
Download
Express in top 10 samples
| Sample | Tissue | Condition | FPKM |
|---|---|---|---|
| SRR1039573 | gastrointestinal | diet 0.75 NPM | 1527.93 |
| SRR1039571 | gastrointestinal | diet 0.1 NPM | 950.48 |
| SRR1534351 | larvae | 500 ug/l BDE47 treatment | 80.88 |
| SRR633550 | larvae | exposed to hypoxia stress | 32.49 |
| SRR942764 | embryo | myd88-/- | 23.44 |
| SRR1342217 | embryo | infection with Mycobacterium marinum | 22.65 |
| SRR519732 | 7 dpf | FETOH treatment | 21.53 |
| SRR519727 | 6 dpf | FETOH treatment | 21.17 |
| SRR1167753 | embryo | bacterial infection | 20.84 |
| SRR1534349 | larvae | DMSO vehicle | 18.92 |
Express in tissues
Correlated coding gene
Download
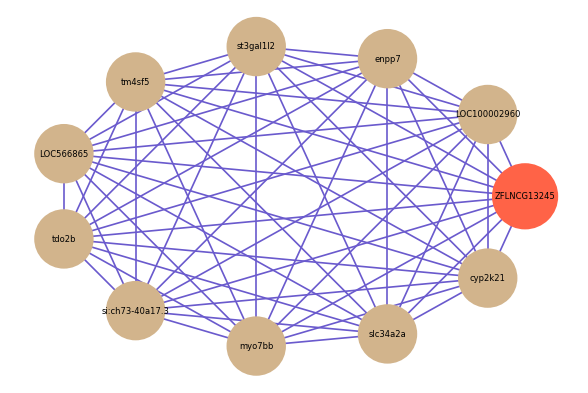
| gene | correlation coefficent |
|---|---|
| LOC100002960 | 0.65 |
| enpp7 | 0.65 |
| st3gal1l2 | 0.65 |
| tm4sf5 | 0.64 |
| LOC566865 | 0.64 |
| tdo2b | 0.63 |
| si:ch73-40a17.3 | 0.63 |
| myo7bb | 0.62 |
| slc34a2a | 0.62 |
| cyp2k21 | 0.62 |
Gene Ontology
Download
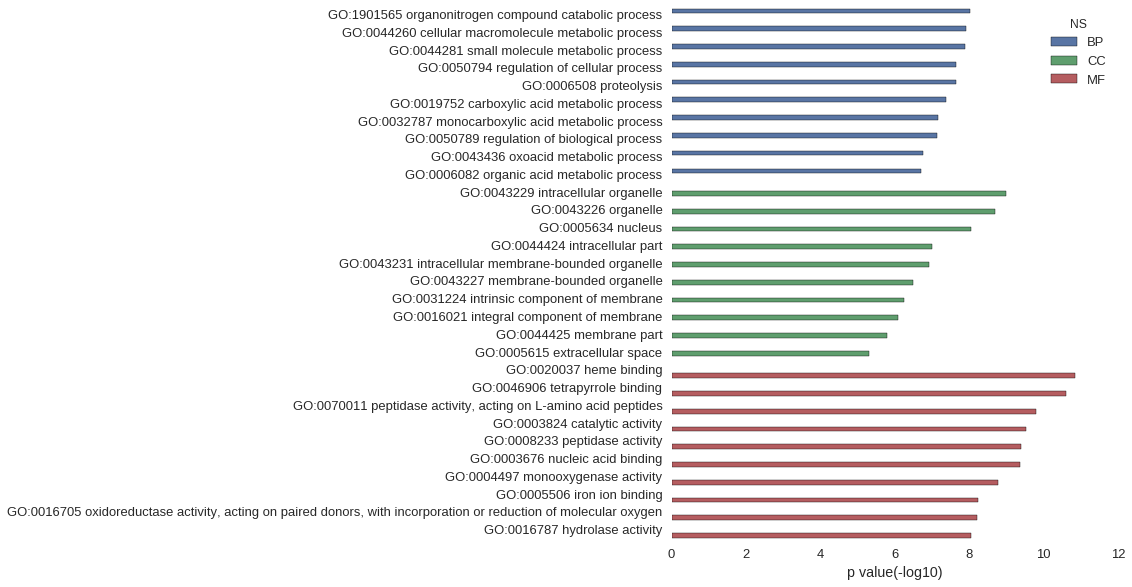
| GO | P value |
|---|---|
| GO:1901565 | 9.29e-09 |
| GO:0044260 | 1.23e-08 |
| GO:0044281 | 1.28e-08 |
| GO:0050794 | 2.27e-08 |
| GO:0006508 | 2.32e-08 |
| GO:0019752 | 4.20e-08 |
| GO:0032787 | 6.84e-08 |
| GO:0050789 | 7.27e-08 |
| GO:0043436 | 1.69e-07 |
| GO:0006082 | 1.93e-07 |
| GO:0043229 | 1.02e-09 |
| GO:0043226 | 2.02e-09 |
| GO:0005634 | 8.65e-09 |
| GO:0044424 | 9.69e-08 |
| GO:0043231 | 1.22e-07 |
| GO:0043227 | 3.21e-07 |
| GO:0031224 | 5.63e-07 |
| GO:0016021 | 8.18e-07 |
| GO:0044425 | 1.57e-06 |
| GO:0005615 | 5.04e-06 |
| GO:0020037 | 1.41e-11 |
| GO:0046906 | 2.44e-11 |
| GO:0070011 | 1.60e-10 |
| GO:0003824 | 3.00e-10 |
| GO:0008233 | 4.02e-10 |
| GO:0003676 | 4.30e-10 |
| GO:0004497 | 1.69e-09 |
| GO:0005506 | 5.67e-09 |
| GO:0016705 | 6.23e-09 |
| GO:0016787 | 9.06e-09 |
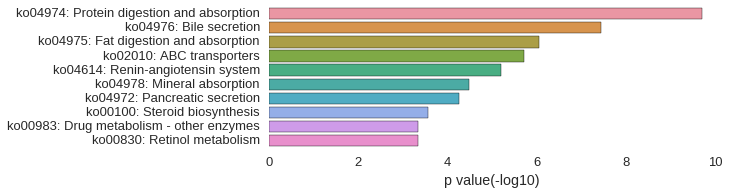
KEGG Pathway
Download
Conservation
OMIM
OMIM of this lncRNA gene hava does not exist!
Sequence Download
>ZFLNCG13245
ACACATTCAAATCTCCTTAACGGGGCATTGTGTTTCCCTAAGAACGCTGCATCAGCTCCTCTGTCAGTAC
TGTGTGATATGAAGCCTCCATTAGCTGATAACGTCTGCTGTTAGTGAACATCAGTGCCGGTAGTGACGTG
TAGGTGTGTGTGTGTGTGTGTTTGTTTTTTCCTCTGTCTTCAGGGTGTGCTGTATGAGATCGATTACTCG
CGTCTGTCCTGCCAGAAGAAGGCTCTAGAGTCGACCTTTCACCCCACGCGGGTGCCGGCTGATGCTGTCT
TCTTAGGGCAGGTGATTCTGGGCACCTCGTCTGTGCCGGGACTCGGGCTCCTCAGCAACTCATGGGTCGG
AGAGATTGCAGAGATACAGGGTCAGTCTACTAACTAGAACTACTGCACTCACTGATGCGAATGAGTAAGG
TGATGGTGAGCTAATAATGACGATGATGATGTAATGGTGAGGATTATGATTTTGGTGATGGTAATGATGT
ACTAATGACAAGGATGAAGATGATGTAATGATTATTATGATGTGATAGTAATGATGTAGATGGTGATGAT
AAAGATGATGGTAATAATGACGTTGATGTAATGATGACGTTGATGGTAATGATGATGTTGATGGTAATGA
TGAAGATGATGTAATGATGATGATGGTAATGATGACGTTGATGGTAATGATGATGTTGATGGTAATGATG
AAGATGATGTAATGATGATGATGGTAATGATGATGGTGATGGTAATGATGATGATGGTAATGATGAAGAT
GATGGTAATGATGATGATGATGGTAATGATGAAGATGATGGTAATGATGATGTTGATGTAATGATGACGT
TGATGGTAATGATGATGTTGATGGTAATGATGAAGATGATGTAATGATGATGATGGTAATGATGATGGTG
ATGGTAATGATGATGATGGTAATGATGAAGATGATGGTAATGATGACGTTGATGGTAATGATGAAGATGA
TGGTAATGATG
ACACATTCAAATCTCCTTAACGGGGCATTGTGTTTCCCTAAGAACGCTGCATCAGCTCCTCTGTCAGTAC
TGTGTGATATGAAGCCTCCATTAGCTGATAACGTCTGCTGTTAGTGAACATCAGTGCCGGTAGTGACGTG
TAGGTGTGTGTGTGTGTGTGTTTGTTTTTTCCTCTGTCTTCAGGGTGTGCTGTATGAGATCGATTACTCG
CGTCTGTCCTGCCAGAAGAAGGCTCTAGAGTCGACCTTTCACCCCACGCGGGTGCCGGCTGATGCTGTCT
TCTTAGGGCAGGTGATTCTGGGCACCTCGTCTGTGCCGGGACTCGGGCTCCTCAGCAACTCATGGGTCGG
AGAGATTGCAGAGATACAGGGTCAGTCTACTAACTAGAACTACTGCACTCACTGATGCGAATGAGTAAGG
TGATGGTGAGCTAATAATGACGATGATGATGTAATGGTGAGGATTATGATTTTGGTGATGGTAATGATGT
ACTAATGACAAGGATGAAGATGATGTAATGATTATTATGATGTGATAGTAATGATGTAGATGGTGATGAT
AAAGATGATGGTAATAATGACGTTGATGTAATGATGACGTTGATGGTAATGATGATGTTGATGGTAATGA
TGAAGATGATGTAATGATGATGATGGTAATGATGACGTTGATGGTAATGATGATGTTGATGGTAATGATG
AAGATGATGTAATGATGATGATGGTAATGATGATGGTGATGGTAATGATGATGATGGTAATGATGAAGAT
GATGGTAATGATGATGATGATGGTAATGATGAAGATGATGGTAATGATGATGTTGATGTAATGATGACGT
TGATGGTAATGATGATGTTGATGGTAATGATGAAGATGATGTAATGATGATGATGGTAATGATGATGGTG
ATGGTAATGATGATGATGGTAATGATGAAGATGATGGTAATGATGACGTTGATGGTAATGATGAAGATGA
TGGTAATGATG